
Biyoinformatik Analiz Hizmetleri
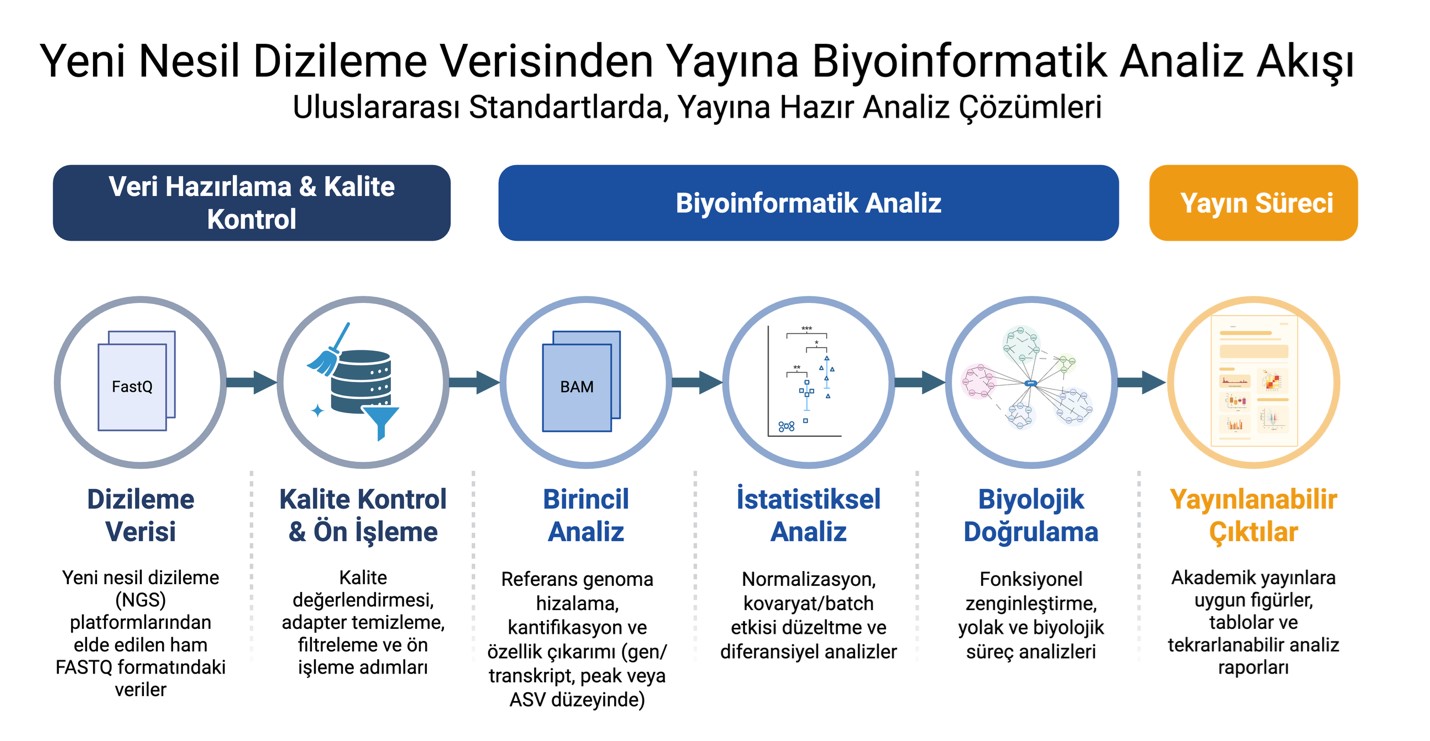
Biyoinformatik Analiz Birimimiz, yüksek hacimli yeni nesil dizileme (NGS) verilerinin biyolojik olarak anlamlı, istatistiksel olarak güvenilir ve yayınlanabilir sonuçlara dönüştürülmesini amaçlayan bir hizmet platformudur. Akademik araştırma grupları ve Ar-Ge ekiplerine yönelik olarak; RNA-seq, single-cell analizler, epigenomik, metagenomik ve tüm genom dizileme (WGS) başta olmak üzere geniş bir analiz yelpazesinde, uluslararası standartlara uygun, şeffaf ve tekrarlanabilir çözümler sunmaktayız.
Genomik, transkriptomik ve proteomik veriler için uçtan uca biyoinformatik analiz hizmetleri sağlayan birimimizde; veri kalite kontrolü ve ön işleme adımlarından ileri düzey istatistiksel analizlere, biyolojik yorumlamadan yayın destek süreçlerine kadar tüm aşamalar bilimsel doğruluk ve metodolojik tutarlılık esas alınarak yürütülmektedir. Projelere özel olarak tasarlanan analiz stratejileri ile araştırma süreçlerinin hızlandırılması ve yüksek güvenilirlikte sonuçlar elde edilmesi hedeflenmektedir.
Hizmetlerimiz
1. RNA-seq / miRNA / tRNA Analizleri
RNA dizileme verilerinin kalite kontrolünden diferansiyel ekspresyon ve fonksiyonel yorumlamaya kadar kapsamlı analizi.
7. Yayın Destek Hizmetleri
Akademik yayınlar için şekil, tablo, metod yazımı ve supplementary materyal desteği.
8. Pipeline Geliştirme (Nextflow)
Tekrar üretilebilir, ölçeklenebilir ve dokümante edilmiş biyoinformatik analiz pipeline’larının geliştirilmesi.
9. Danışmanlık & Deney Tasarımı
Deney planlaması, analiz stratejisi ve veri yorumlama süreçlerinde birebir bilimsel danışmanlık.
Katkı Sağladığımız Yayınlar
Öztemur Islakoğlu, Y., Korhan, P., Binokay, L., Keleş, B., Bağırsakçı, E., Uludağ Taşçıoğlu, M., Şamdancı, E., Karakülah, G., & Atabey, N. (2025). Fusion transcripts landscape in hepatocellular carcinoma and potential impact on the expression of fusion partners. RNA Biology, 22(1), 1–16. https://doi.org/10.1080/15476286.2025.2529036
Karacicek, B., Katkat, E., Binokay, L., Ozhan, G., Karakülah, G., & Genc, S. (2025). The role of tRNA fragments on neurogenesis alteration by H₂O₂-induced oxidative stress. Journal of Molecular Neuroscience, 75(2), 1–12. https://doi.org/10.1007/s12031-025-02330-x
Arioz, B. I., Binokay, L., Tastan, B., Genc, B., Cotuk, A., Dursun, E., Gezen-Ak, D., Hanagası, H., Gurvit, İ. H., Bilgic, B., Bagriyanik, A., Karakülah, G., Yener, G. G., & Genc, S. (2025). Characterization of tRNA-derived fragments in the small neuron-derived extracellular vesicles of Alzheimer’s disease patients. Brain Research, 1862, 149730. https://doi.org/10.1016/j.brainres.2025.149730
Günay, Ç., Binokay, L., Karakülah, G., Polat, İ., Yiş, U., & Hiz-Kurul, S. (2025). Exploring molecular pathways underlying epilepsy development in intellectual disability. Neuropediatrics.
Karabicici, M., Akbari, S., Caliskan, C., Celiker, C., Oz, O., Binokay, L., Karakülah, G., Senturk, S., & Erdal, E. (2025). Modeling hepatic fibrosis in TP53 knockout iPSC-derived human liver organoids. Molecular Oncology. https://doi.org/10.1002/1878-0261.70119
Koçak, G., Uyulgan, S., Polatlı, E., Sarı, V., Kahveci, B., Bursalı, A., Binokay, L., Reçber, T., Nemutlu, E., Mardinoğlu, A., Karakülah, G., Utine, C. A., & Güven, S. (2024). Generation of anterior segment of the eye cells from hiPSCs in microfluidic platforms. Advanced Biology, 8(5), 2400018. https://doi.org/10.1002/adbi.202400018
Binokay, L., Oktay, Y., & Karakülah, G. (2024). An API for dynamic estimation of reference intervals for functional abundances of gut microbiota. Biologia, 79(1), 343–353. https://doi.org/10.1007/s11756-023-01556-7
Kahveci, B., Polatlı, E., Evranos, A. E., Güner, H., Baştanlar, Y., Karakülah, G., & Güven, S. (2025). BrAIn: A comprehensive artificial intelligence-based morphology analysis system for brain organoids and neuroscience. bioRxiv. https://doi.org/10.1101/2025.02.19.638973
Vasilopoulos, S. N., Güner, H., Uça Apaydın, M., Pavlopoulou, A., & Georgakilas, A. G. (2023). Dual targeting of DNA damage response proteins implicated in cancer radioresistance. Genes, 14(12), 2227. https://doi.org/10.3390/genes14122227https://doi.org/10.3390/genes14122227
Gunalp, S., Goksu Helvaci, D., Oner, A., Bursalı, A., Conforte, A., Güner, H., Karakülah, G., Szegezdi, E., & Sag, D. (2023). TRAIL promotes the polarization of human macrophages toward a proinflammatory M1 phenotype and is associated with increased survival in cancer patients with high tumor macrophage content. Frontiers in Immunology, 14, 1209249. https://doi.org/10.3389/fimmu.2023.1209249
Birim Sorumlusu: Leman BİNOKAY
