Hesaplamalı Yapısal Biyoloji
Baş Araştırmacı: Dr. Öğr. Üyesi Ezgi KARACA
Karaca Lab, biyomoleküler komplekslerin yapı-fonksiyon ilişkilerini hesaplamalı yaklaşımlar kullanarak anlamaya odaklanmaktadır. Deneysel yöntemler pek çok biyomoleküler yapının çözülmesini sağlamış olsa da, genomik, proteomik ve biyokimya gibi alanlardaki veri artış hızına yetişmekte yetersiz kalmaktadır. Bu bilgi açığını kapatmak amacıyla, Yapısal Biyoloji alanında en güncel hesaplamalı araçları hem kullanıyor hem de geliştiriyoruz. Son yıllarda yapay zeka (YZ) uygulamalarının çığır açan başarısıyla, YZ tabanlı yapısal tahmin araçlarını iş akışımıza entegre ederek omik verileri yapısal düzeyde daha etkili bir şekilde yorumluyoruz.
Araştırmalarımız, biyomoleküler etkileşimleri ve mekanizmaları ortaya koyacak yüksek doğrulukta yapısal modeller üretmeyi hedeflemektedir. Gelişmiş modelleme tekniklerini biyolojik verilerle harmanlayarak, proteinlerin moleküler düzeyde işleyişini daha derinlemesine anlamaya katkıda bulunuyoruz.
Araştırma Alanları
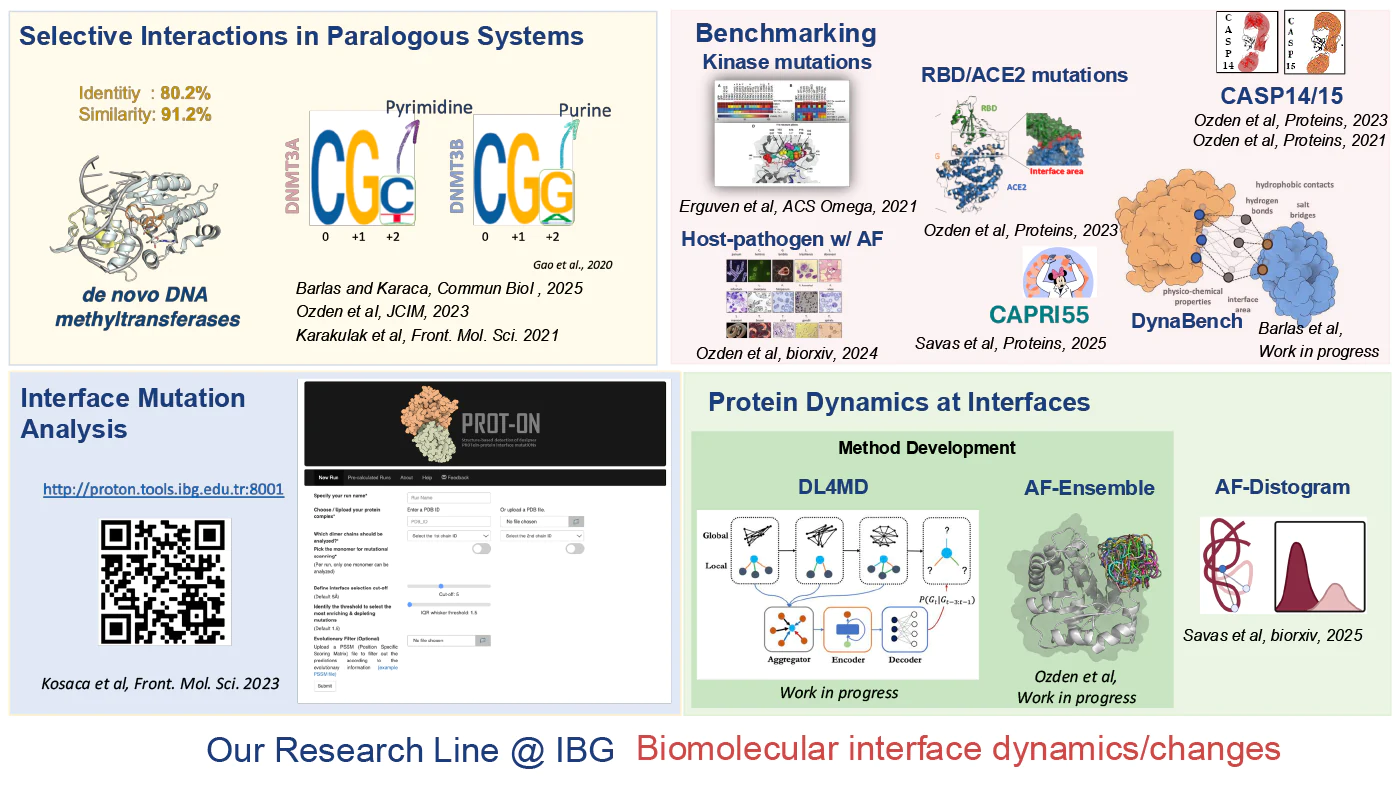
Paralog Sistemlerde Seçici Etkileşimler: De novo DNA metiltransferazlar gibi yüksek dizi ve yapı benzerliği gösteren proteinler arasındaki özgül etkileşim mekanizmalarını keşfetmek için ileri düzey yapısal modelleme yaklaşımları ve moleküler dinamik simülasyonları kullanarak protein–protein arayüzlerindeki özgüllük belirleyici faktörleri analiz edilmektedir.
Arayüz Mutasyonlarının Etkileri: Protein-protein etkileşim yüzeylerindeki mutasyonların yapısal etkileri araştırılmaktadır. Bu kapsamda geliştirilen PROT-ON (http://proton.tools.ibg.edu.tr:8001/result/dae4ee4df0ec47e9a119fd512fdbfd98) adlı web sunucumuz ile arayüz mutasyonlarının etkileri interaktif olarak incelenebilmektedir.
YZ ve Fizik Tabanlı Modelleme Yaklaşımları ile Arayüz Karakterizasyonu and Karşılaştırması: Kinaz mutasyonları, RBD–ACE2 ve konak–patojen etkileşimleri gibi sistemlerin arayüzleri, YZ ve fizik tabanlı modelleme yaklaşımlarıyla modellenmektedir ve bu yöntemlerin doğruluk ve başarı düzeylerini karşılaştırmalı olarak değerlendirilmektedir.
Arayüz Özelliklerine Dayalı Değerlendirme Araçlarının Geliştirilmesi: Protein arayüzlerinin yapısal ve fizikokimyasal özelliklerini detaylı şekilde analiz edebilen, Python tabanlı DynaBench analiz paketi geliştirilmektedir.
Moleküler Dinamikleri Anlamak için Yöntem Geliştirme: Protein arayüzlerinin dinamiklerini modellemek için DL4MD ve AF-Ensemble projelerimizle YZ temelli yöntem geliştirme çalışmalarımız devam etmektedir.
YZ Tabanlı Kompleks Protein Yapı Tahmini: Protein–protein komplekslerinin yapısal tahmini için yeni nesil düşük kaynak tüketimli YZ yaklaşımı, MinnieFold (https://github.com/CSB-KaracaLab/MinnieFold), geliştirilmektedir. Yaklaşımımızın başarısı CAPRI yarışmasının 55. turunda test edilmiştir.
Protein Kompleks Modelleme Yaklaşımlarının Değerlendirilmesi: Yapısal biyoloji alanındaki güncel modelleme yöntemlerinin doğruluk ve uygulanabilirliğini değerlendirmek amacıyla, CASP Assembly kategorisinde; CASP14 ve CASP15 dönemlerinde hakemlik görevleri gerçekleştirilmiştir. Bu kapsamda, protein komplekslerinin tahminine yönelik yaklaşımlar bağımsız, sistematik ve objektif metriklerle analiz edilmiştir.
Yayınlar
Schildhauer, F., Ryl, P. S., Lauer, S. M., Lenz, S., Barlas, A. B., Ouzounidis, V. R., Jeffrey, K., Marcu, D., O’Reilly, F. J., Graziadei, A., Stuiver, M., Schmidt, K., Ewers, H., Spahn, C. M. T., Karaca, E., ... & Rappsilber, J. An NADH-controlled gatekeeper of ATP synthase. Molecular Cell. 2025.
Savaş, B., Yılmazbilek, İ., Özsan, A., & Karaca, E. Towards a Greener AlphaFold2 Protocol for Antibody–Antigen Modeling: Insights From CAPRI Round 55. Proteins: Structure, Function, and Bioinformatics. 2025.
Honorato, R. V., Trellet, M. E., Jiménez-García, B., Schaarschmidt, J. J., Giulini, M., Reys, V., Koukos, P. I., Rodrigues, J. P. G. L. M., Karaca, E., ... & Bonvin, A. M. The HADDOCK2. 4 web server for integrative modeling of biomolecular complexes. Nature protocols, 19(11), 3219-3241. 2024.
Ozden, B., Şamiloğlu, E., Özsan, A., Erguven, M., Yükrük, C., Koşaca, M., ... & Karaca, E.. Benchmarking the accuracy of structure‐based binding affinity predictors on Spike–ACE2 deep mutational interaction set. Proteins: Structure, Function, and Bioinformatics, 92(4), 529-539. 2024.
Ozden, B., Kryshtafovych, A., & Karaca, E.. The impact of AI‐based modeling on the accuracy of protein assembly prediction: insights from CASP15. Proteins: Structure, Function, and Bioinformatics, 91(12), 1636-1657. 2023.
Projeler
EMBO Installation Grant #4421: Identification of Pioneer Transcription Factor Binding Modes to Chromatin
TÜBİTAK 2509 Türkiye - Fransa Bosphorus ikili işbirliği çağrısı: Exploring AlphaFold for Generation of Conformational Ensembles of Protein Complexes (AF-Ensemble)
Ekip
Aktif Üyeler
Ayşe Berçin Barlas (Doktora Sonrası Araştırmacı)
Burcu Özden (Doktora Sonrası Araştırmacı)
Büşra Savaş (Doktora)
İbrahim Atakan Kubilay (Doktora)
Buse Şahin, (Yüksek Lisans)
Murat Taylan Şahin (Yüksek Lisans)
Gülce Berfin Ercan (Lisans)
Önceki Üyeler
Ayşe Berçin Barlas, (Doktora)
Burcu Özden, (Doktora)
Mehdi Koşaca, (Yüksek Lisans)
Eda Şamiloğlu, (Yüksek Lisans)
Deniz Doğan, (Yüksek Lisans)
Tülay Karakulak, (Yüksek Lisans)